Resumos
A colibacilose é a enfermidade entérica de maior impacto na suinocultura, sendo ocasionada por cepas enterotoxigênicas de Escherichia coli. Quarenta isolados clínicos de suínos com diarréia e 13 isolados ambientais foram analisados quanto ao perfil genotípico, relação genética e resistência antimicrobiana. O gene que codifica para a toxina Stb foi identificado em 50% dos isolados clínicos, seguido por Sta e Lt, com 35%. Dentre os fatores de adesinas pesquisados, a F18 foi encontrada em 27,5% das amostras. A técnica de ERIC-PCR utilizada para caracterização epidemiológica dos isolados, não demonstrou poder discriminatório esperado, e apesar de permitir a separação dos isolados em grupos, estes não evidenciaram grupos relacionados aos fatores de virulência. No teste de susceptibilidade antimicrobiana a maior resistência foi observada à tetraciclina, em 88,6%. O índice de resistência múltipla aos antimicrobianos (IRMA), variou entre 0 a 0,69.
Escherichia coli; perfil genotípico; resistência
Colibacillosis is an enteric disease with a major impact to the swine industry and is caused by enterotoxigenic strains of Escherichia coli. Forty clinical isolates from pigs with diarrhea and 13 environmental isolates were analysed regarding their genotypic profile, genetic relationship and antibiotic resistance. The most prevalent gene was Stb, identified in 50% of the isolates from clinical cases, and Sta and Lt were detected in 35% of them. Among the adesine factors investigated, F18 was found in 27.5% of the E. coli strains. The ERIC-PCR technique used for epidemiological characterization of the isolates did not show the expected discriminatory power. However, the test allowed separation of the isolates in groups, but did not evidence groups related to virulence factors. In the susceptibility test, the highest values for resistance were to tetracycline, in 88.6%. The index of multiple resistance to antimicrobials varied from 0 to 0.69.
Escherichia coli; genotypic profile; resistance
Caracterização epidemiológica, molecular e perfil de resistência aos antimicrobianos de Escherichia coli isoladas de criatórios suínos do sul do Brasil
Epidemiology, molecular characterization and resistance to antimicrobials of Escherichia coli isolated from South-Brazilian pig herds
Mateus Matiuzzi da CostaI; Mariana Sá e SilvaII; Denis Augusto SpricigoIII; Niura Mazzini WittII; Silvana Beutinger MarchioroII; Lilian KollingI; Agueda Palmira Castagna de VargasII, * * Autor para correspondência. E-mail: agueda@ccr.ufsm.br O arquivo disponível sofreu correções conforme ERRATA publicada no Volume 26 Número 2 da revista.
IPrograma de Pós-Graduação em Biologia Celular e Molecular, Universidade Federal do Rio Grande do Sul (UFRGS); Universidade do Oeste de Santa Catarina (UNOESC), Campus Xanxerê, SC
IIDepto Medicina Veterinária Preventiva, Laboratório de Bacteriologia, Universidade Federal de Santa Maria (UFSM), prédio 44, sala 5147, Santa Maria, RS 97105-900
IIIPrograma de Pós-Graduação em Medicina Veterinária, UDESC, Lages, SC
RESUMO
A colibacilose é a enfermidade entérica de maior impacto na suinocultura, sendo ocasionada por cepas enterotoxigênicas de Escherichia coli. Quarenta isolados clínicos de suínos com diarréia e 13 isolados ambientais foram analisados quanto ao perfil genotípico, relação genética e resistência antimicrobiana. O gene que codifica para a toxina Stb foi identificado em 50% dos isolados clínicos, seguido por Sta e Lt, com 35%. Dentre os fatores de adesinas pesquisados, a F18 foi encontrada em 27,5% das amostras. A técnica de ERIC-PCR utilizada para caracterização epidemiológica dos isolados, não demonstrou poder discriminatório esperado, e apesar de permitir a separação dos isolados em grupos, estes não evidenciaram grupos relacionados aos fatores de virulência. No teste de susceptibilidade antimicrobiana a maior resistência foi observada à tetraciclina, em 88,6%. O índice de resistência múltipla aos antimicrobianos (IRMA), variou entre 0 a 0,69.
Termos de indexação:Escherichia coli, perfil genotípico, resistência.
ABSTRACT
Colibacillosis is an enteric disease with a major impact to the swine industry and is caused by enterotoxigenic strains of Escherichia coli. Forty clinical isolates from pigs with diarrhea and 13 environmental isolates were analysed regarding their genotypic profile, genetic relationship and antibiotic resistance. The most prevalent gene was Stb, identified in 50% of the isolates from clinical cases, and Sta and Lt were detected in 35% of them. Among the adesine factors investigated, F18 was found in 27.5% of the E. coli strains. The ERIC-PCR technique used for epidemiological characterization of the isolates did not show the expected discriminatory power. However, the test allowed separation of the isolates in groups, but did not evidence groups related to virulence factors. In the susceptibility test, the highest values for resistance were to tetracycline, in 88.6%. The index of multiple resistance to antimicrobials varied from 0 to 0.69.
Index terms:Escherichia coli, genotypic profile, resistance.
INTRODUÇÃO
A colibacilose é a enfermidade entérica de maior impacto na suinocultura, sendo ocasionada por cepas enterotoxigênicas de Escherichia coli (ETEC). Para o desenvolvimento da doença as bactérias aderem-se à mucosa intestinal e produzem uma ou mais enterotoxinas (Lt, Sta e Stb), que levam ao desenvolvimento de diarréia e desidratação, podendo resultar na morte dos animais. Os tipos de adesinas comumente associados com a doença são K88 (F4), K99 (F5), 987P (F6) e F41 (Dean-Nystrom et al. 1997, Bertschinger & Fairbrother 1999).
Somente o isolamento de E. coli a partir de amostras de fezes e conteúdo intestinal não é suficiente para o diagnóstico da enfermidade. Nesse sentido, as técnicas de soroaglutinação, ELISA, imunofluorescência e PCR (genotipificação) podem ser empregadas para a biotipificação de adesinas e toxinas (Wills 2000).
A técnica de ERIC (Enterobacterial repetitive intergenic consensus) é baseada na análise de seqüências cromossomais repetidas, que tem sido usada para a caracterização clonal de diferentes espécies de enterobactérias e para o estudo da relação genética entre isolados. Entretanto, o ERIC-PCR tem sido utilizado com êxito para a diferenciação clonal de cepas de Shigella sonnei, Salmonella typhimurium e Vibrio cholerae (Versalovic et al. 1991, Rivera et al. 1995), e no estudo de variabilidade genética de E. coli patogênicas para aves (Silveira et al. 2002).
O uso de fármacos antimicrobianos para o controle e profilaxia de enfermidades bacterianas em suínos é amplamente difundido. Porém, essa prática está associada à presença de resíduos de antibióticos nos alimentos de origem animal e seleção de bactérias resistentes, trazendo sérios riscos à saúde pública (Dunlop et al. 1998).
Os objetivos deste estudo foram investigar o perfil genotípico através da PCR e o índice de resistência em amostras de E. coli provenientes de isolados clínicos e ambientais, além de caracterizar epidemiologicamente os isolados de E. coli, determinando uma relação genética entre os isolados pelo uso do ERIC-PCR e da genotipificação.
MATERIAL E MÉTODOS
Cepas bacterianas
Quarenta isolados clínicos de suínos provenientes de granjas da região sul do Brasil e 13 isolados ambientais foram utilizados neste trabalho. Os isolados clínicos foram obtidos de fezes, intestino delgado e fígado de suínos com diarréia, remetidos ao laboratório de Bacteriologia da UFSM. Os isolados ambientais foram obtidos de duas amostras de água do bebedouro, uma da esterqueira, três de fezes de animais sadios, um de inseto, um de swab da parede da granja, três de swabs do piso e duas da ração ofertada aos animais. As amostras ambientais foram coletadas em uma granja na cidade de Coronel Freitas, Santa Catarina.
Os isolados foram semeados em meios de cultura ágar MacConkey (MC) e ágar sangue (AS). As colônias suspeitas foram identificadas por suas características morfológicas, bioquímicas, tintoriais, e presença de hemólise, conforme Quinn et al. (1994). Uma colônia isolada das culturas positivas foi repicada para ágar sangue e utilizada na caracterização molecular.
Caracterização genotípica
O DNA das colônias caracterizadas fenotipicamente como Escherichia coli foi termo-extraído e o sobrenadante resultante foi utilizado como DNA molde da reação em cadeia da polimerase (PCR). A PCR multiplex foi realizada com 35 ciclos de amplificação compostos de 30 segundos a 94ºC, 30 segundos a 55ºC e 90 segundos a 72ºC, e uma extensão final de 10 min a 72ºC. Nove fatores de virulência foram pesquisados, sendo Sta, Stb, Lt e Stx as toxinas e F4, F5, F41, F6 e F18 as adesinas, sintetizados segundo Dean-Nystrom (2004). Os fragmentos utilizados como controles dos iniciadores podem ser observados na Fig.1.
Caracterização epidemiológica
A técnica de rep-PCR foi realizada com 24 amostras clínicas de diferentes regiões. O DNA genômico foi amplificado por PCR (40 ciclos de 30 segundos a 94ºC, 60 segundos a 37ºC, e 90 segundos a 72ºC, com extensão final de 10 min a 72ºC), utilizando como iniciadores as seqüências ERIC 1 (5' - ATG TAA CT CCT GGG GAT TCA C - 3') e ERIC 2 (5' - AAG TAA GTG ACT GGG GTG AGC G - 3') descritos por Manke et al. (1998).
Teste de susceptibilidade aos antimicrobianos
O teste foi realizado pelo método de difusão em disco Kirby-Bauer modificado. Os antimicrobianos testados foram: ampicilina (10µg), cefalexina (30µg), enrofloxacina (15µg), colistina (10µg), norfloxacina (10µg), fluorfenicol (30µg), amicacina (30µg), cloranfenicol (30µg), trimetoprim (05µg), neomicina (30µg), tetraciclina (30µg), sulfazotrim (sulfametoxazol + trimetoprim) (25µg) e gentamicina (10µg).
O índice de resistência múltipla aos antimicrobianos (IRMA) foi calculado conforme metodologia descrita por Kruperman (1983), sendo este índice determinado pela relação entre o número de antimicrobianos que a amostra é resistente e o número total de antimicrobianos testados.
RESULTADOS E DISCUSSÃO
A hemólise em AS foi verificada em 85% (34/40) dos isolados clínicos, e apenas em 7,6% (1/13) das amostras ambientais, concordando com a observação de que a maior parte das cepas patogênicas de Escherichia coli isoladas de suínos são hemolíticas, ao passo que para as outras espécies domésticas esta característica não é observada (Henton & Hunter 1994). A hemólise é, portanto, utilizada como indicativo de virulência em isolados de suínos, sendo muito útil na caracterização fenotípica.
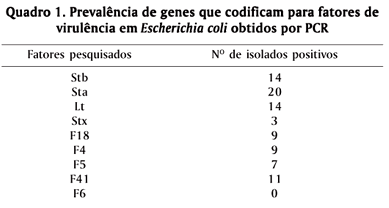
Os dados obtidos na PCR podem ser observados no Quadro 1. Dos 40 isolados clínicos, 82,5% (33/40) foram positivos para pelo menos um dos genes pesquisados que codificam fatores de virulência. Os genes para toxinas mais prevalentes foram de Stb, presentes em 50% (20/40) dos isolados, seguidos de Sta e Lt com 35%. Os genes de adesinas com maior prevalência foram F18, com 27,5%, seguido por F4 e F5 com 22,5%. Resultados semelhantes para a prevalência das toxinas foram obtidos por Moon et al. (2004), onde Stb e Lt foram as toxinas de maior freqüência. Entretanto, o mesmo autor encontrou a fímbria F4 em maior freqüência, seguida pela F18. A toxina STb é normalmente encontrada nos isolados de suínos, entretanto é rara nos isolados obtidos de outras espécies como o ser humano (Casey et al. 1998). Os mesmos autores reportam que esta toxina sozinha causa a perda de fluídos nos intestinos de leitões inoculados, contudo não produz os efeitos severos da diarréia característica de animais infectados em condições naturais. O presente estudo revelou dois isolados positivos somente para Stb e F41. Contudo, não se pode predizer uma menor patogenicidade a estes isolados, pois muitas vezes a presença de infecções concomitantes, em especial as víricas, pode acentuar a patogenicidade de isolados de ETEC (Nakamine et al. 1998).
Nenhum isolado foi positivo para o gene de F6, dado divergente do observado por Martins et al. (2000), que encontrou 35,7% de amostras positivas para este fator. O mesmo autor também encontrou índice de 1,2% para F5, muito abaixo do observado neste experimento, onde o índice foi de 22,5%. Este fato pode ser atribuído a diferenças entre as populações estudadas.
O gene que codifica para Stx foi identificado em 7,5% (3/40) das amostras. Algumas cepas produtoras da toxina Stx de E. coli podem causar diarréia, colite hemorrágica e a síndrome urêmica hemolítica em humanos. A presença destes genes em amostras oriundas de animais tem sido alvo de preocupação, já que existe a probabilidade da infecção humana e o suíno ser incriminado neste processo (Oswald et al. 2000).
Genes que codificam para fatores de virulência têm sido encontrados em amostras ambientais ou de animais sadios de E. coli isoladas no Brasil (Martins et al. 2000), sugerindo o potencial patogênico independente de estarem causando doença clínica. Este dado não foi confirmado no presente trabalho, já que todas as amostras ambientais foram negativas para a presença dos genes pesquisados.
Os dados obtidos com a utilização da técnica de rep-PCR permitiram a construção de um dendrograma de similaridade (dado não publicado). Apesar de a técnica permitir a separação dos isolados em grupos, estes não demonstraram a correlação esperada com a patogenicidade, semelhante ao observado por Silveira et al. (2001), que não observaram relação de patogenicidade em cepas de E. coli isoladas de humanos com infecção do trato urinário. A técnica também demonstrou baixo poder discriminatório quando utilizada para a pesquisa de relação genética de isolados de E. coli de suínos com diarréia pós desmame (Osek 2000).
Dentre os isolados analisados, 98,1% (52/53) apresentaram resistência a pelo menos um dos agentes antimicrobianos testados, sendo que a única amostra com sensibilidade a todos os antimicrobianos foi isolada da ração ofertada aos animais. A multi-resistência tem sido citada por diversos autores (Baccaro et al. 2002, Guerra et al. 2003) apresentando níveis mais elevados em cepas isoladas de suínos e aves quando comparada a isolados de bovino (Guerra et al. 2003), fato que se deve à pressão de seleção causada pela adição indiscriminada de antibióticos na ração animal.
Amostras de E. coli usualmente são sensíveis a gentamicina, amicacina, trimetoprim, sulfazotrim (sulfametoxazol + trimetoprim) e ceftiofur, e resistentes a tetraciclina, estreptomicina, sulfonamidas, ampicilina e kanamicina (Hirsh & Zee 1999), dados não confirmados nesta pesquisa, onde o maior índice de sensibilidade foi observado para fluorfenicol, em 81,1% (43/53) dos isolados seguido de norfloxacina com 79,2% e enrofloxacina, com 75,4%. A maior resistência foi observada para tetraciclina em 88,6% (47/53), seguida por sulfazotrim 73,5% e por trimetoprim, com 66% (Fig.2). A divergência observada pode ser atribuída à variabilidade das cepas e ao crescente aumento da resistência antimicrobiana entre as enterobactérias.
O estudo de Baccaro et al. (2002), em amostras de E. coli isoladas de fezes de leitões de granjas no estado de São Paulo, revelou resistência de 87,4% das amostras a sulfazotrim, e 86,8% a ampicilina, dados semelhantes aos observados nesta pesquisa, porém o mesmo autor encontrou resistência de 92% à norfloxacina, enquanto neste estudo foi observada a resistência de apenas 15% a este antimicrobiano. A divergência de dados pode ser atribuída ao fato dos autores citados terem utilizado a concentração inibitória mínima (CIM) como metodologia na determinação da resistência, e também a variação regional.
O índice de resistência múltipla aos antimicrobianos (IRMA), variou entre 0 e 0,69. Este índice foi desenvolvido para o estudo do risco de contaminação de alimentos por isolados de E. coli, e serve como uma informação adicional do potencial patogênico das amostras (Kruperman 1983).
Os resultados desta pesquisa demonstraram que a maior parte dos isolados clínicos possuem genes que codificam para os fatores de virulência pesquisados, tendo a adesina F18 e a toxina Stb como os fatores de maior prevalência, evidenciando que os isolados de Escherichia coli na região sul do Brasil possuem grande potencial patogênico. Foi também observada a grande variabilidade genética, porém a técnica de ERIC-PCR não mostrou eficiência na discriminação dos isolados.
Evidencia-se uma alta resistência das amostras estudadas à tetraciclina, sulfazotrim (sulfametoxazol + trimetoprim) e trimetoprim. O alto índice de resistência múltipla observado tem aumentado consideravelmente, e um dos principais fatores é o uso excessivo de antimicrobianos. Porém um correto manejo sanitário, profilaxia e higiene são formas simples de prevenção de doenças, podendo contribuir para a redução da resistência bacteriana.
Agradecimentos.- Os autores agradecem a FAPERGS pelo suporte financeiro (ARD 01/1633.9 e 02/60063.8).
Recebido em 17 de janeiro de 2005.
Aceito para publicação em 23 de julho de 2005.
- Baccaro M.R., Moreno A.M., Corrêa A., Ferreira A.J.P. & Calderaro F.F. 2002. Resistência antimicrobiana de amostras de Escherichia coli isoladas de fezes de leitões com diarréia. Arqs Inst. Biológico, São Paulo, 69(2):15-18.
- Bertschinger H.U. & Fairbrother J.M. 1999. Escherichia coli infections, p.431-468. In: Straw B.E., D'allaire S., Mengeling W.L. & Taylor, D.J. (ed.) Diseases of Swine. Iowa State University Press, Ames.
- Casey T.A., Herring C.J., Schneider R.A., Bosworth B.T. & Whipp S. 1998. Expression of heat stable enterotoxin STb by adherent Escherichia coli is not sufficient to cause severe diarrhea in neonatal pigs. Infect. Immun. 66: 1270-1272.
- Dean-Nystrom E.A. 2004. Personal communication (USDA, ARS, National Animal Disease Center, P.O. Box 70, Ames, IA 50010-0070, USA. E-mail: enystrom@nadc.ars.usda.gov).
- Dean-Nystrom E.A., Burkhardt D., Bosworth B.T. & Welter M.W. 1997. Presence of F18ac (2134 P) fimbriae on 4 P-E.coli isolates from weaned pigs with diarrhea. Vet. Clin. North America, Food Anim. Practice 9:77-79.
- Dunlop R.H., McEwen S.A., Meek A.H., Clarke R.C., Black W.D. & Friendship R.M. 1998. Associations among antimicrobial drug treatments and antimicrobial resistance of fecal E. coli swine on 34 farrow-to-finish farms in Ontario, Canada. Prev. Vet. Med. 34:283-305.
- Guerra B., Junker E., Schoroeter A., Malorny B., Lehmann S. & Helmuth R. 2003. Phenotypic and genotypic characterization of antimicrobial resistance in German Escherichia coli isolates from cattle, swine and poultry. J. Antimicrob. Chemother. 52:489-492.
- Hirsh D.C. & Zee Y.C. 1999. Veterinary Microbiology. Blackwell Science, Malden, p.69-74.
- Henton M.M. & Hunter P. 1994. E. coli infections, p.1085-1099. In: Coetzer J.A.W., Thomsom G.R., Tustin R.C. (ed.) Infectious Diseases of Livestock with special reference to Southern Africa. Oxford University Press, Cape Town.
- Krumperman P.H. 1983. Multiple antibiotic resistance indexing of Escherichia coli to identify high-risk sources of fecal contamination of foods. Appl. Environ. Microbiol. 46(1):165-170.
- Manke T., Wesley I.V., Dickson J.S. & Harmon K.M. 1998. Prevalence and genetic variability of Arcobacter species in mechanically separated turkey. J. Food Prot. 61(12):1623-1628.
- Martins M.F., Rossi-Martinez N.M., Ferreira A., Brochi M., Yano T., Castro A.F.P. & Silveira W.D. 2000. Pathogenic characteristics of Escherichia coli strains isolated from newborn piglets with diarrhea in Brazil. Vet. Microbiol. 76:51-59.
- Moon H.W., Booher S.L., Cornick N.A. & Hoffman, L.J. 2004. Prevalence of virulence factors among Escherichia coli Iowa State University Press, Ames. Disponível em: http://www.extension.iastate.edu/Pages/ansci/swinereports/asl-1601.pdf Acesso em 3 de maio 2004.
- Nakamine M., Kono Y., Abe S., Hoshino C., Shirai J. & Ezaki T. 1998. Dual infection with enterotoxigenic Escherichia coli and porcine reproductive and respiratory syndrome virus, observed in weaning pigs that died suddenly. J. Vet. Med. Sci. 60: 555-561.
- Osek J. 2000. Virulence factors and genetic relatedness of Escherichia coli strains isolated from pigs with post-weaning diarrhea. Vet. Microbiol. 71:211-222.
- Oswald E., Schimdt H., Morabito S., Karch H., Marches O. & Caprioli A. 2000. Typing of intimin human and animal enterohemorragic and enteropathogenic Escherichia coli: Characterization of a new intimin variant. Infect.Immun. 68:64-71.
- Quinn P.J., Carter M.E. & Markey B. 1994. Clinical Veterinary Microbiology. Wolfe, London, p.209-236.
- Rivera I.G., Chowdhury M.A.R., Huq A., Jacobs D., Martius M.T. & Colwell R.R. 1995. Enterobacterial repetitive intergenic consensus sequences and the PCR to generate fingerprints of genomic DNAs from Vibrio cholerae O1, O139 and non-O1 strains. Appl. Environ. Microbiol. 61:2898-2904.
- Silveira W.D., Benetti F., Lancellotti M., Ferreira A., Solferini V.N. & Brocchi M. 2001. Biological and genetic characteristics of uropathogenic Escherichia coli strains. Revta Inst. Med. Trop., São Paulo, 43(6):303-310.
- Silveira W.D., Ferreira A., Lancellotti M., Barbosa I.A., Leite D.S., Castro A.F. & Brocchi M. 2002. Clonal relationships among avian Escherichia coli isolates determined by enterobacterial repetitive intergenic consensus (ERIC)-PCR. Vet. Microbiol. 89:323-328.
- Versalovic J., Koeuth T. & Lupski J.R. 1991. Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes. Nucleic Acids Res. 19:6823-6831.
- Wills R.W. 2000. Diarrhea in growing-finishing swine. Vet. Clin. North America, Food Anim. Practice 16(1):175-185.
Datas de Publicação
-
Publicação nesta coleção
16 Out 2006 -
Data do Fascículo
Mar 2006
Histórico
-
Recebido
17 Jan 2005 -
Aceito
23 Jul 2005