Resumos
A cultura da cana-de-açúcar é de extrema importância no cenário agrícola nacional. No entanto, pouco se sabe sobre a estruturação das comunidades microbianas associadas aos solos e às rizosferas de tais plantas. O objetivo deste trabalho foi avaliar a estrutura e diversidade das comunidades de bactérias associadas ao solo e à rizosfera de seis variedades de cana-de-açúcar cultivadas no Estado de São Paulo (Brasil). As análises foram realizadas com base em métodos independentes de cultivo, em que a técnica de PCR-DGGE revelou alterações na rizosfera para os grupos de bactérias totais e também para os grupos de Alphaproteobacteria e Betaproteobacteria. Após essa análise, quatro amostras (três de rizosfera e uma de solo) foram usadas para o sequenciamento da região V6 do gene 16S DNAr na plataforma Ion Torrent TM. Essa análise gerou um total de 95.812 sequências, dentro das quais houve a predominância das afiliadas aos filos Actinobacteria, Proteobacteria e Acidobateria . Os resultados revelaram que as comunidades bacterianas na rizosfera são distintas daquelas encontradas no solo. Foi possível ainda observar efeito diferencial de plantas das variedades. Alguns grupos bacterianos apresentaram menor frequência na rizosfera (Acidobacteria ), enquanto outros se mostraram fortemente estimulados pela presença das raízes, comumente para todas as variedades (Betaproteobacteria , Nitrospora e Chloroflexi ), ou em respostas variedade-específicas (Bacilli e Sphingobacteria ).
métodos independentes de cultivo; genes ribossomais; diversidade bacteriana; sequenciamento de DNA
Sugarcane is an extremely important crop in Brazilian agriculture. However, little is known about the structure of microbial communities associated with the soils and rhizosphere of these plants. The aim of this study was to evaluate the structure and diversity of bacterial communities associated with the soil and rhizosphere of six sugarcane varieties grown in the State of São Paulo, Brazil. Analyses were carried out based on methods not dependent on the crop, where the PCR-DGGE technique revealed alterations in the rhizosphere for the total bacteria group, and also for the Alphaproteobacteria and Betaproteobacteria groups. After that analysis, four samples (three from the rhizosphere and one from the soil) were subjected to sequencing of the V6 region of the 16S rDNA gene using the IonTorrentTM platform. This approach generated 95,812 sequences, with the prevalence of those affiliated with the phyla Actinobacteria , Proteobacteria , and Acidobacteria . Results revealed that bacterial communities in the rhizosphere are distinct from those observed in soils. It was also possible to observe a differential selection performed by plants of different varieties. Some bacterial groups exhibited lower frequencies in the rhizosphere (Acidobacteria ), while others proved to be highly stimulated by the presence of roots, in a response common to all varieties (Betaproteobacteria , Nitrospora , and Chloroflexi ), or in variety-specific responses (Bacilli and Sphingobacteria ).
cultivation-independent methods; ribosomal genes; bacterial diversity; DNA sequencing
DIVISÃO 2 - PROCESSOS E PROPRIEDADES DO SOLO
COMISSÃO 2.1 - BIOLOGIA DO SOLO
Composição diferencial das comunidades bacterianas na rizosfera de variedas de cana-de-açúcar1 1 Trabalho extraído da Dissertação de Mestrado do primeiro autor.
Differential composition of bacterial communities in the rhizosphere of sugarcane varieties
Diogo Paes da CostaI; Armando Cavalcante Franco DiasII; Ademir DurrerI; Pedro Avelino Maia de AndradeI; Thiago GumiereI; Fernando Dini AndreoteIII
IDoutorando, Departamento de Ciência do Solo, Laboratório de Microbiologia. ESALQ/USP. E-mail: diogocosta1@yahoo.com.br, demadurrer@yahoo.com.br, pedro890@hotmail.com, thiago.gumiere@gmail.com
IIPós-doutorando, Departamento de Ciência do Solo, Laboratório de Microbiologia, ESALQ/USP. E-mail: dias147@gmail.com
IIIProfessor, Departamento de Ciência do Solo, Escola Superior de Agricultura "Luiz de Queiroz" - ESALQ/USP. Av. Pádua Dias, 11. Caixa Postal 09. CEP 13418-900 Piracicaba (SP), Brasil. E-mail: fdandreo@gmail.com
RESUMO
A cultura da cana-de-açúcar é de extrema importância no cenário agrícola nacional. No entanto, pouco se sabe sobre a estruturação das comunidades microbianas associadas aos solos e às rizosferas de tais plantas. O objetivo deste trabalho foi avaliar a estrutura e diversidade das comunidades de bactérias associadas ao solo e à rizosfera de seis variedades de cana-de-açúcar cultivadas no Estado de São Paulo (Brasil). As análises foram realizadas com base em métodos independentes de cultivo, em que a técnica de PCR-DGGE revelou alterações na rizosfera para os grupos de bactérias totais e também para os grupos de Alphaproteobacteria e Betaproteobacteria. Após essa análise, quatro amostras (três de rizosfera e uma de solo) foram usadas para o sequenciamento da região V6 do gene 16S DNAr na plataforma Ion Torrent TM. Essa análise gerou um total de 95.812 sequências, dentro das quais houve a predominância das afiliadas aos filos Actinobacteria, Proteobacteria e Acidobateria . Os resultados revelaram que as comunidades bacterianas na rizosfera são distintas daquelas encontradas no solo. Foi possível ainda observar efeito diferencial de plantas das variedades. Alguns grupos bacterianos apresentaram menor frequência na rizosfera (Acidobacteria ), enquanto outros se mostraram fortemente estimulados pela presença das raízes, comumente para todas as variedades (Betaproteobacteria , Nitrospora e Chloroflexi ), ou em respostas variedade-específicas (Bacilli e Sphingobacteria ).
Termos de indexação: métodos independentes de cultivo, genes ribossomais, diversidade bacteriana, sequenciamento de DNA
ABSTRACT
Sugarcane is an extremely important crop in Brazilian agriculture. However, little is known about the structure of microbial communities associated with the soils and rhizosphere of these plants. The aim of this study was to evaluate the structure and diversity of bacterial communities associated with the soil and rhizosphere of six sugarcane varieties grown in the State of São Paulo, Brazil. Analyses were carried out based on methods not dependent on the crop, where the PCR-DGGE technique revealed alterations in the rhizosphere for the total bacteria group, and also for the Alphaproteobacteria and Betaproteobacteria groups. After that analysis, four samples (three from the rhizosphere and one from the soil) were subjected to sequencing of the V6 region of the 16S rDNA gene using the IonTorrentTM platform. This approach generated 95,812 sequences, with the prevalence of those affiliated with the phyla Actinobacteria , Proteobacteria , and Acidobacteria . Results revealed that bacterial communities in the rhizosphere are distinct from those observed in soils. It was also possible to observe a differential selection performed by plants of different varieties. Some bacterial groups exhibited lower frequencies in the rhizosphere (Acidobacteria ), while others proved to be highly stimulated by the presence of roots, in a response common to all varieties (Betaproteobacteria , Nitrospora , and Chloroflexi ), or in variety-specific responses (Bacilli and Sphingobacteria ).
Index terms: cultivation-independent methods, ribosomal genes, bacterial diversity, DNA sequencing
INTRODUÇÃO
A cana-de-açúcar (Sacchharum spp.) é uma das culturas mais importantes do mundo, representada por cerca de 30 espécies pertencentes à família Poaceae , que são cultivadas principalmente nas regiões tropicais (Mendes et al., 2007; Dini-Andreote et al., 2010). O Brasil é atualmente o maior produtor mundial dessa cultura, ocupando aproximadamente 8,35 milhões de ha, contando com produção de cerca de 560,36 milhões de t e rendimento médio de 67,060 t ha-1 na safra 2011/2012 (CONAB, 2012).
Vários trabalhos têm dado especial atenção à estrutura das comunidades microbianas associadas à cana-de-açúcar, buscando a promoção de crescimento vegetal e a aplicação biotecnológica por meio dos processos de fixação biológica de N, solubilização de fosfato inorgânico, antagonismo a fitopatógenos, reguladores de crescimento vegetal e produção de enzimas de interesse industrial, entre outros (Makoi et al., 2008; Compant et al., 2010; Luvizotto et al., 2010; Mendes et al., 2011).
As regiões mais próximas às raízes da cana-de-açúcar, principalmente o rizoplano e a rizosfera, são os locais preferenciais de colonização de microrganismos, sendo essa predileção relacionada à sinalização molecular entre plantas e microrganismos (por exemplo, intermediada por flavonoides ou isoflavonoides) ou em razão da grande disponibilidade de nutrientes oriundos da exsudação radicular, contribuindo com o metabolismo e a multiplicação de células microbianas (Baldani et al., 1997; Medeiros et al., 2006, Drigo et al., 2010). Esse fato tem dado suporte ao desenvolvimento de trabalhos sobre a diversidade microbiana da rizosfera (Cocking, 2003; Kang & Mills, 2004; Carvalho et al., 2010; Philippot et al., 2013).
Apesar da grande diversidade de microrganismos associados à cana-de-açúcar, apenas uma pequena fração é conhecida e pode ser cultivada, apontando a limitação dos métodos tradicionais de bioprospecção da biodiversidade microbiana nos ambientes complexos (Amann et al., 1995). Nesse aspecto, as técnicas independentes de cultivo têm contribuído para o conhecimento da diversidade e do potencial biotecnológico das espécies microbianas (Marques et al., 2008; Andreote et al., 2009; Lee & Wong, 2009).
As combinações de diferentes métodos de estudo têm fornecido valiosas informações sobre a diversidade, a funcionalidade e as interações que ocorrem na colonização microbiana da rizosfera (Dumont et al., 2011). Esses métodos são eficientes quanto ao fornecimento de dados mais refinados, que podem ser estudados por técnicas mais comuns de análises independentes de cultivo, como a DGGE, a biblioteca de clones ou até mesmo o sequenciamento, feitos com base em genes filogenéticos ou funcionais, dando suporte a inferências sobre o papel de determinados organismos em processos específicos no ambiente em que se encontram (Huang et al., 2009).
Diante do exposto, este trabalho foi realizado objetivando avaliar as alterações entre as estruturas das comunidades de bactérias nativas do solo e da rizosfera de diferentes variedades de cana-de-açúcar cultivadas em distintas regiões do Estado de São Paulo, permitindo melhor compreensão da composição e diversidade de bactérias associadas ao solo e à rizosfera da cana-de-açúcar.
MATERIAL E MÉTODOS
O experimento foi realizado com seis variedades de cana-de-açúcar: RB85-5156, RB85-5453, RB86-7515, PO8862, SP5541 e SP5543. Essas variedades foram coletadas de canteiros da Fazenda Areão, situada no município de Piracicaba, SP (22º 41,716' S, 47º 38,478' O), estando todas no mesmo estádio fisiológico. As amostras foram coletadas em triplicatas, mantendo-se distância mínima de aproximadamente 50 m de uma amostragem para outra, coletando-se tanto as plantas quanto o solo em volta das raízes em cada ponto amostral. As raízes foram envoltas em sacos plásticos para a preservação da umidade, tomando-se cuidado em manter boa porção de solo agregado. As plantas coletadas foram levadas para o laboratório, onde foi feita a separação do solo e da rizosfera. As porções não aderidas às raízes foram utilizadas para compor as amostras de solo, enquanto a fração do solo ligada às estruturas radiculares das plantas, a uma distância máxima de 2,0 mm, foi removida cuidadosamente para compor as amostras rizosféricas. Dessa forma, foram obtidas 18 amostras de rizosfera e 18 amostras de solo, totalizando 36 amostras. Amostras da rizosfera receberam o prefixo 'R' enquanto as do solo receberam o prefixo 'S' na nomenclatura.
As amostras obtidas de solo e rizosfera foram pesadas em frações padronizadas de 0,4 g, as quais foram usadas para extração de DNA, realizada com kit comercial PowersoilTM DNA Isolation kit (MoBio Laboratories Inc, Carlsbad, CA, EUA ), de acordo com instruções do fabricante.
As amostras de DNA foram usadas na análise de eletroforese em gel de gradiente desnaturante (DGGE), realizada para o grupo de bactérias totais de acordo com método descrito por Heuer et al. (1997), com os iniciadores U968GC e 1387R. Além da análise de bactérias totais, foram feitas as análises de DGGE para os grupos de Alphaproteobacteria e Betaproteobacteria , seguindo o método descrito por Gomes et al. (2001), utilizando-se os iniciadores AlphaU e Beta-2 como seletivos numa amplificação inicial, seguida de uma nested PCR, com os iniciadores U968GC e 1387R. Após obter os fragmentos, os produtos da PCR foram avaliados em gel de agarose a 1 %, seguido de coloração em brometo de etídeo e visualização em luz ultravioleta. A análise de DGGE foi realizada no aparelho phorU2 system (Ingeny, Goes, Holanda). O gradiente desnaturante (ureia e formamida deionizada) foi de 45 a 65 %, formados a partir de soluções-estoque de poliacrilamida (6 %) contendo 0 e 100 % de desnaturantes. As condições da eletroforese foram de 65 ºC por 16 h a 100 V. Após a eletroforese, os géis foram corados com SYBR-gold (Invitrogen, Breda, The Netherlands) em TAE 0,5x no escuro por 120 min. As imagens resultantes foram convertidas em matrizes de presença/ausência de bandas com o software ImageQuant TL unidimensional (Amersham Biosciences, Amersham, UK, v.2003) (McCaig et al., 2001).
O software PAST 1.90 (Hammer et al., 2001) foi utilizado para a análise dos dados binários e agrupamentos por similaridade dos perfis, com base no algoritmo UPGMA (Unweighted Pair-Group Method with Arithmetical Average ) e no coeficiente de similaridade de Bray-Curtis. Para avaliar a variação entre os grupos encontrados na análise de cluster , foi empregada a análise de similaridade, que permite testar a ocorrência de diferença significativa entre dois ou mais grupos de amostras (Clarke, 1993). Esse método compara a distância entre os grupos, gerando valores de R que variam de -1 a +1, em que valores próximos a 0 indicam a semelhança entre os grupos, e valores próximos a 1, a perfeita separação desses (Clarke & Gorley, 2006).
As amostras mais representativas, com maior diferenciação da rizosfera em relação ao solo adjacente, de acordo com os resultados obtidos pela análise dos perfis gerados pelo método de DGGE, foram submetidas ao sequenciamento de amplicons na plataforma Ion TorrentTM (Life Technologies), realizado na Embrapa Meio Ambiente, em Jaguariúna, SP. Essa análise foi realizada para três amostras de rizosfera (variedades RB86-7515, RB85-5453 e RB85-5156) e uma de solo, usado neste caso o coletado próximo ao cultivo da variedade RB86-7515. Para tanto, o DNA extraído foi amplificado com iniciadores franqueadores da região V6 do gene 16S DNAr descritos por Sogin et al. (2006). Amplicons dessas amostras foram gerados com iniciadores marcados com distintas tags , formadas por cinco pares de bases (barcodes ), permitindo a identificação da origem das sequências geradas (Vamps, 2012).
Para análise das sequências obtidas, inicialmente foi utilizado o programa CLC Genomics Workbench (CLCbio) , usado para remover as sequências de baixa qualidade e para separar as sequências de cada amostra com base nos barcodes . Em seguida, as análises foram realizadas utilizando o programa QIIME (Quantitative Insights Into Microbial Ecology) (Caporaso et al., 2010), em que foram agrupadas em OTU (Operational Taxonomic Units ), compostas de sequências com similaridade mínima de 97 %. Os números de OTU, e suas respectivas frequências, foram computados para o cálculo dos índices de diversidade de Shannon e para a estimativa de riqueza de espécies, pelo método não paramétrico de Chao1 (Chao & Bunge, 2002).
A afiliação taxonômica das sequências foi realizada por comparação com as sequências presentes no RDP (Ribosomal Database Project ), em que foram analisadas com base na ferramenta Classifier (Cole et al., 2007).
RESULTADOS E DISCUSSÃO
A técnica de PCR-DGGE permitiu inferir sobre a estrutura das comunidades microbianas presente nas diferentes amostras. Os dendrogramas obtidos com base nos resultados de tais análises evidenciaram de forma robusta o agrupamento das repetições, indicando claramente uma separação entre as comunidades bacterianas do solo e da rizosfera para as diferentes variedades de cana-de-açúcar (Figura 1). Ainda, pode-se observar maior similaridade entre as amostras de solo, enquanto as amostras da rizosfera de cada variedade se apresentaram mais distintas entre si (Figura 1). Essa observação dá suporte à literatura, indicando que plantas diferentes selecionam diferentes grupos de microrganismos na sua rizosfera a partir da mesma microbiota do solo. Destaca-se que no momento da amostragem todas as plantas apresentavam-se no mesmo estádio fenológico, o que elimina variações decorrentes deste fator. As diferenças existentes entre as estruturas das comunidades de bactérias do solo e da rizosfera ocorreram por causa da diferenciação na quantidade e composição dos exsudatos radiculares, determinados por diferenças genotípicas entre as plantas (Kang & Mills, 2004; Medeiros et al., 2006; Compant et al., 2010). Essas diferenças certamente resultam no metabolismo diferencial das variedades, causando alterações na composição e quantidade de material liberado nas raízes. Maior conhecimento sobre esse tipo de efeito, em que a alteração do genótipo da planta leva a diferenciações na estrutura da comunidade microbiana da rizosfera, é de extrema importância para o melhoramento genético que considere a engenharia da rizosfera como fator de seleção. Esse fato foi comprovado por Andreote et al. (2008), os quais verificaram o efeito da alteração genética e metabólica de plantas de tabaco na composição da comunidade microbiana da rizosfera, durante o período de maior atividade radicular de tais plantas (período vegetativo em comparação ao período de senescência). Essa observação indica que a exsudação radicular da planta pode transpor a pressão de seleção exercida por outros fatores como tipo de solo e condições ambientais, na estruturação da comunidade microbiana da rizosfera.
Os resultados deste estudo são condizentes com os perfis obtidos para a análise de bactérias totais com os perfis alcançados para as análises de Alphaproteobacteria , observando claramente a diferenciação das amostras de rizosfera oriundas do solo (Figura 1), o que indicou que a maioria dos grupos microbianos, entre eles o composto pelas Alphaproteobacteria , é selecionada na rizosfera de cana-de-açúcar. Combinando essa informação com as constantes no quadro 3, verificou-se que a alteração na estrutura dessa comunidade não necessariamente reflete numa maior quantidade relativa de sequências desse grupo nas amostras da rizosfera, sendo a seleção com base na estruturação da comunidade e não na magnitude dela. Numa mesma percepção, a comunidade de Betaproteobacteria evidencia-se mais similar entre as amostras de solo e rizosfera, possivelmente indicando menor seleção de grupos pertencentes a essa classe no ambiente rizosférico. Nascimento (2006), estudando estrutura das comunidades de Betaproteobacteria , observou que as variações em tais grupos estavam mais relacionadas à frequência relativa do que com à diversidade, o que pode indicar que o parâmetro usado neste estudo não seja o melhor para detectar efeitos rizosféricos sobre o grupo das Betaproteobacteria .
No intuito de validar as separações observadas, a análise de ANOSIM foi realizada, demonstrando que em todas as frações analisadas da comunidade existe uma separação das amostras de rizosfera e do solo; porém, essas apresentam grupos microbianos em comum (Quadro 1). A análise de similaridade foi feita também para comparar a diferença nas estruturas de bactérias da rizosfera das diferentes variedades de cana-de-açúcar, revelando distinção entre todas as amostras, e para todas as frações das comunidades analisadas (bactérias totais, Alphaproteobacteria e Betaproteobacteria ), resultando em valores de RBray-Curtis = 1,000 (p<0,01).
Esses resultados permitiram a seleção de algumas amostras representativas de rizosfera (selecionando as três variedades com resultados mais distinto no PCR-DGGE) e uma amostra de solo (em razão da maior similaridade entre as amostras) para uma análise mais detalhada, realizada por meio de sequenciamento parcial do gene ribossomal na plataforma IonTorrent TM. Essa análise gerou um conjunto de 95.812 sequências (em média 23.953 por amostra) com um fragmento de aproximadamente 75 pb. Utilizando o software QIIME, foi possível a afiliação das sequências às OTU, a 97 % de similaridade, e a posterior estimativa dos índices de riqueza e diversidade. Essa análise indicou maior similaridade nos valores obtidos para as amostras de rizosfera do que para aqueles observados na única amostra de solo analisada. Essa observação complementou os resultados de DGGE, indicando que, apesar de distintas, as amostras de rizosfera preservaram maior similaridade em sua composição taxonômica do que a observada quando essas são confrontadas com a amostra de solo. Foi observado que tanto a riqueza como a diversidade apresentaram valores maiores nas amostras de rizosfera (Quadro 2). Esse é um fato controverso, uma vez que, se a planta recruta organismos para compor sua rizosfera, essa será composta por uma fração das bactérias já presente no solo (Philippot et al., 2013). A detecção de maiores valores de diversidade e riqueza na rizosfera pode estar associada ao método empregado, visto que o sequenciamento de genes ribossomais não amostra todos os componentes da comunidade, e o número diferente de sequências analisadas dentro de cada uma das amostras pode levar a algum bias de análise. Além disso, limitações podem ainda ser encontradas no kit de extração de DNA, sendo esse conhecidamente pouco eficaz em representar grupos compostos por uma pequena quantidade de células (espécies raras). Na amplificação do material, tem-se também a amplificação preferencial de fragmentos mais frequentes (espécies abundantes). Apesar de esses fatores serem conhecidos, não existe forma de realizar uma análise livre dessas interferências. Dessa forma, essa discussão serve para justificar o fato de maiores valores de riqueza e diversidade terem sido encontrados nas amostras de rizosfera deste trabalho. Também, um fator que pode contribuir para essa observação é a possibilidade de a planta atuar como fonte de inóculo microbiano em sua rizosfera. Nessa percepção, tem-se que a ocorrência de maior riqueza de espécies nesse ambiente seria relacionado à sua colonização por organismos endofíticos da planta, que passariam a ocupar esse nicho, como descrito por Patriquin & Döbereiner (1978).
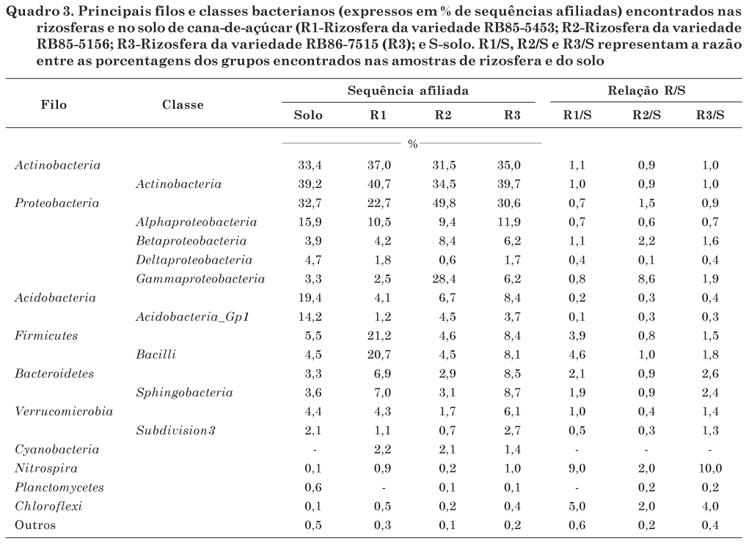
A análise taxonômica permitiu a classificação das sequências geradas, sendo 88 % dessas alocadas dentro de grupos bacterianos já descritos. Os filos mais abundantes encontrados foram: Actinobacteria, Proteobacteria, Acidobacteria, Firmicutes, Bacteriodetes e Verrucomicrobia, seguidos de filos menos abundantes, como Cyanobacteria, Nitrospira, Planctomycetes e Cloroflexi (Quadro 3). A maioria desses grupos foi também encontrada como a mais abundante em outro estudo com solos de cana-de-açúcar (Dini-Andreote et al., 2010); entretanto, nesta pesquisa, são adicionados os grupos Bacteriodetes e Verrucomicrobia , possivelmente não detectados no trabalho anterior em razão do método empregado (no trabalho de Dini-Andreote, o levantamento foi feito com clones, e o número de sequências analisadas é muito menor - total de 193 sequências). A presença de tais organismos no solo é amplamente discutida, e várias justificativas são apresentadas para isso, como a participação da microbiota do solo na decomposição da matéria orgânica (McCaig et al., 2001), o que é amplamente atribuído a Proteobacteria em áreas de pastagem (Nüsslein & Tiedje, 1999) ou à interação com plantas, como descrito para o grupo das Actinobacteria , frequentemente relacionado a gramíneas (Dunbar et al., 2002).
No entanto, o foco deste trabalho esteve na seleção exercida pela planta sobre tal comunidade, em que os dados mostraram, por meio da determinação das relações R/S, comportamento diferenciado dos grupos microbianos detectados (Quadro 3). A primeira observação que chamou a atenção foi o valor positivo dessa relação para o grupo Betaproteobacteria , que corroborou o observado nas análises de DGGE na literatura (Nascimento, 2006), sendo o efeito rizosférico sobre esse grupo mais relacionado à sua frequência relativa do que à composição da comunidade. O contrário foi observado para Alphaproteobacteria , que demonstrou alteração na estrutura de sua comunidade por DGGE, porém associado a baixos valores de relação R/S (Figura 1, Quadro 3). Nessa análise, observou-se ainda pequeno efeito da rizosfera no incremento da porcentagem de sequências classificadas dentro do filo Actinobacteria (relações R/S entre 0,9 e 1,1), ao passo que outros grupos revelaram-se responsivos à presença das raízes. O grupo das Acidobacteria apresentou-se pouco atraído pela presença de raízes, tendo sua comunidade mais abundante na amostra de solo (valores de R/S entre 0,2 e 0,4). Em contrapartida, incrementos, em termos percentuais, de determinadas comunidades foram observados. Esse incremento foi observado de forma homogênea entre as variedades apenas para grupos menos abundantes, como Nitrospira e Chloroflexi , com relações R/S variando entre 2,0 a 10,0 e 2,0 a 5,0, respetivamente (Quadro 3). Pouco se sabe sobre o papel desses grupos microbianos no solo e, ou, associados às plantas, sendo esse importante alvo em estudos posteriores. Já outros grupos majoritários demonstraram resposta diferencial de acordo com o genótipo da planta. As rizosferas de plantas das variedades RB85-5453 e RB86-7515 apresentaram razões R/S mais similares do que as observadas para a rizosfera de plantas da variedade RB85-5156. Essa observação pode ser feita, por exemplo, porque o grupo de Gammaproteobacteria teve valor R/S maior para plantas RB85-5156 (8,6 contra 0,8 e 1,9), enquanto os valores maiores de R/S foram observados para os outros dois genótipos para os grupos Bacilli (4,6 e 1,8 contra 1,0) e Sphingobacteria (1,9 e 2,4 contra 0,9) (Quadro 3). Isso sugeriu que enquanto algumas plantas selecionaram grupos bacterianos relacionados à Gammaproteobacteria , outras apresentam maior eficiência na promoção do desenvolvimento de bactérias relacionadas às classes Bacilli e Sphingobacteria .
As variações observadas na estrutura das comunidades bacterianas da rizosfera e do solo de cana-de-açúcar foram claras, podendo ocorrer por diversos motivos. O papel da presença das raízes sobre propriedades físicas e químicas do solo é amplamente conhecido, sendo essas também fonte de formas assimiláveis de nutrientes, com destaque para o C (Cerri et al., 2004; Philippot et al., 2013). No entanto, essa seleção também é dependente da dinâmica e plasticidade dos organismos que compõem a microbiota dos solos, elevando a capacidade adaptativa frente às alterações ambientais (Abby & Daubim, 2007). Podem ser descritos como fatores diferenciais entre o solo e a rizosfera os teores de C lábil (Araújo et al., 2011), a intensidade da competição entre microrganismos (Fontaine et al., 2007), e a exsudação radicular (Carney et al., 2007; Drigo, et al., 2010; Jia & Zhou, 2011). Neste trabalho, adicionou-se a este ambiente complexo o efeito de variedades distintas, sendo essas capazes de selecionar grupos distintos de bactérias do solo para compor suas respetivas rizosferas.
De forma geral, este trabalho apontou que a cana-de-açúcar estabelece um nicho específico na sua zona rizosférica, tornando essa comunidade diferenciada daquela observada no solo. Nesse contexto, o estudo da diversidade microbiana associada a plantas de cana-de-açúcar é importante, buscando a identificação dos principais grupos responsivos à presença das plantas e, posteriormente, visando melhor exploração do recurso microbiano onipresente nos solos agrícolas.
CONCLUSÕES
1. As comunidades bacterianas da rizosfera e do solo de cana-de-açúcar nas variedades estudadas foram distintas em sua composição.
2. No local amostrado, os filos de Actinobacteria , Proteobacteria e Acidobacteria foram os principais encontrados no solo e na rizosfera de cana-de-açúcar.
3. Os grupos mais responsivos à rizosfera foram Betaproteobacteria , Nitrospora , Chloroflexi (incrementados na rizosfera); Acidobacteria (prevalente no solo); Bacilli e Sphingobacteria (com respostas específicas para distintas variedades).
AGRADECIMENTOS
À Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP), pelo apoio financeiro (processo 2011/03487-2) e pela concessão de bolsa de estudo (processo 2011/04857-8). Aos Dr. Itamar Soares de Melo e Dr. Rodrigo Gouvêa Taketani, pelo suporte no sequenciamento do material na plataforma IonTorrent .
LITERATURA CITADA
Recebido em 20 de fevereiro de 2014 e aprovado em 5 de setembro de 2014
- ABBY, S. & DAUBIN, V. Comparative genomics and the evolution of prokaryotes. Trends Microbiol., 3:135-141, 2007.
- AMANN, R.I.; LUDWIG, W. & SCHELIEFER, K.H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiol. Rev., 59:143-169, 1995.
- ANDREOTE, F.D.; MENDES, R.; DINI-ANDREOTE, F.; ROSSETTO, P.B.; LABATE, C.A.; PIZZIRANI-KLEINER, A.A.; van ELSAS, J.D.; AZEVEDO, J.L. & ARAÚJO, W.L. Transgenic tobacco revealing altered bacterial diversity in the rhizosphere during early plant development. Ant. Leeuw., 93:415-424, 2008.
- ANDREOTE, F.D.; AZEVEDO, J.L. & ARAÚJO, W.L. Assessing the diversity of bacterial communities associated with plants. Braz. J. Microbiol., 40:417-432, 2009.
- ARAÚJO, E.A.; KER, J.C.; MENDONÇA, E.S.; SILVA, I.R. & OLIVEIRA, E.K. Impacto da conversão floresta - pastagem nos estoques e na dinâmica do carbono e substâncias húmicas do solo no bioma Amazônico. Acta Amaz., 41:103-114, 2011.
- BALDANI, J.I.; CARUSO, L.; BALDANI, V.L.D.; GOI, S.R. & DOBEREINER, J. Recent advances in BNF with non-legume plants. Soil Biol. Biochem., 29:911-922, 1997.
- CAPORASO, J.G.; KUCZYNSKI, J.; STOMBAUGH, J.; BITTINGER, K.; BUSHMAN, F.D.; COSTELLO, E.K.; FIERER, N.; PEÑA, A.G.; GOODRICH, J.K.; GORDON, J.I.; HUTTLEY, G.A.; KELLEY, S.T.; CAVALEIROS, D.; KOENIG, J.E.; LEY, R.E.; LOZUPONE, C.A.; MCDONALD, D.; MUEGGE, B.D.; PIRRUNG, M.; REEDER, J. SEVINSKY, J.R.; TURNBAUGH, P.J.; WALTERS, W.A.; WIDMANN, J.; YATSUNENKO, T.; ZANEVELD, J.R. & KNIGHT, R. QIIME allows analysis of high-throughput community sequencing data. Nat. Meth., 7:335-336, 2010.
- CARNEY, K.M.; HUNGATE, B.A.; DRAKE, B.G. & MEGONIGAL, J.P. Altered soil microbial community at elevated CO2 leads to loss of soil carbon. PNAS, 104:4990-4995, 2007.
- CARVALHO, J.L.N.; AVANZI, J.C.; SILVA, M.L.N.; MELLO, C.R. & CERRI, C.E.P. Potencial de sequestro de carbono em diferentes biomas do Brasil: Uma revisão de literatura. R. Bras. Ci. Solo, 34:277-289, 2010.
- CERRI, C.C.; CERRI, C.E.P.; DAVIDSON, E.A.; BERNOUX, M. & FELLER, C. A ciência do solo e o sequestro de carbono. B. Inf. Soc. Bras. Ci. Solo, 29:29-34, 2004.
- CHAO, A. & BUNGE, J. Estimating the number of species in a stochastic abundance model. Biometrics, 58:531-539, 2002.
- CLARKE, K.R. Non-parametric multivariate analyses of changes in community structure. Aust. J. Ecol., 18:117-143, 1993.
- CLARKE, K.R. & GORLEY, R.N. Primer v6: User manual/tutorial. Plymouth, Primer-E, 2006. 192p.
- COCKING, E. Endophytic colonization of plant roots by nitrogen-fixing bacteria. Plant Soil, 252:169-175, 2003.
- COLE, J.R.; CHAI, B.; FARRIS, R.J.; WANG, Q.; KULAM-SYED-MOHIDEEN, A.S.; MCGARRELL, D.M.; BANDELA, A.M.; CARDENAS, E.; GARRITY, G.M. & TIEDJE, J.M. The ribosomal database project (RDP-II): Introducing my RDP space and quality controlled public data. Acids Nucl. Res., 35:169-172, 2007.
- COMPANT, S.; CLÉMENT, C. & SESSITSCH, A. Plant growth-promoting bacteria in the rhizosphere and endosphere of plants: Their role, colonization, mechanisms involved and prospects for utilization. Soil Biol. Biochem., 42:669-678, 2010.
- COMPANHIA NACIONAL DE ABASTECIMENTO - CONAB. Acompanhamento de safra brasileira (cana-de-açúcar), Brasília, 2012. 18p. (terceiro levantamento)
- DINI-ANDREOTE, F.; ANDREOTE, F. D.; COSTA, R.; TAKETANI, R. G.; VAN ELSAS, J.D. & ARAÚJO, W.L. Bacterial soil community in a Brazilian sugarcane field. Plant Soil, 336:337-349, 2010.
- DRIGO, B.; PIJL, A.S.; DUYTS, H.; KIELAK, A.; GAMPER, H.A.; HOUTEKAMER, M.J.; BOSCHKER, H.T.S.; BODELIER, P.L.E.; WHITELEY, A.S.; van VEEN, J.A. & KOWALCHUK, G.A. Shifting carbon flow from roots into associated microbial communities in response to elevated atmospheric CO2 PNAS, 107:10938-10942, 2010.
- DUMONT, M.G.; POMMERENKE, B.; CASPER, P. & CONRAD, R. DNA-, rRNA- and mRNA-based stable isotope probing of aerobic methanotrophs in lake sediment. Environ. Microbiol., 13:1462-2920, 2011.
- DUNBAR, J.; BARNS, S.M.; TICKNOR, L.O. & KUSKE, C.R. Empirical and theoretical bacterial diversity in four Arizona soils. Appl. Environ. Microbiol., 68:3035-3045, 2002.
- FONTAINE, S.; BAROT, S.; BARRÉ, P.; BDIOUI, N.; MARY, B. & CORNELIA, R. Stability of organic carbon in deep soil layers controlled by fresh carbon supply. Nature, 450:277-280, 2007.
- GOMES, N.C.M.; HEUER, H.; SCHONFELD, J.; COSTA, R.; MENDONCA-HAGLER, L. & SMALLA, K. Bacterial diversity of the rhizosphere of maize (Zea mays ) grown in tropical soil studied by temperature gradient gel electrophoresis. Plant Soil, 232:167-180, 2001.
- HAMMER, Ø.; HARPER, D.A.T. & RYAN, P.D. PAST: Paleontological statistics software package for education and data analysis. Palaeontol. Elect., 4:1-9, 2001.
- HEUER, H.; KRSEK, M.; BAKER, P.; SMALLA, K. & WELLINGTON, E.M.H. Analysis of actinomycete communities by specific amplification of genes encoding 16S rRNA and gel-electrophoretic separation in denaturing gradients. Appl. Environ. Microbiol., 63:3233-3241, 1997.
- HUANG, W.E.; FERGUSON, A.; SINGER, A.C.; LAWSON, K.; THOMPSON, I.P.; KALIN, R.M.; LARKIN, M.J.; BAILEY, M.J. & WHITELEY, A.S. Resolving genetic functions within microbial populations: In situ analyses using rRNA and mRNA stable isotope probing coupled with single-cell Raman-fluorescence in situ hybridization. Appl. Environ. Microbiol., 75:234-241, 2009.
- JIA, X. & ZHOU, C.J. Effects of long-term elevated CO2 on rhizosphere and bulk soil bacterial community structure in Pinus sylvestriformis seedlings fields. Adv. Mater. Res., 351:343-344, 2011.
- KANG, S. & MILLS, A.L. Soils bacterial community structure changes following disturbance of the overlying plant community. Soil Sci., 169:55-65, 2004.
- LEE, A. & WONG, E. Optimization and the robustness of BOX A1R PCR for DNA fingerprinting using trout lake E. coli isolates. J. Exp. Microbiol. Immunol., 13:104-11, 2009.
- LUVIZOTTO, D.M.; MARCON, J.; ANDREOTE, F.; DINI-ANDREOTE, F.; NEVES, A.A.C.; ARAÚJO, W.L. & PIZZIRANI-KLEINER, A.A. Genetic diversity and plant-growth related features of Burkholderia spp. from sugarcane roots. World J. Microbiol. Biotechem., 26:1829-1836, 2010.
- MAKOI, J. & NDAKIDEMI, P. Selected soil enzymes: Examples of their potential roles in the ecosystem. Afric. J. Biotechem., 7:181-191, 2008.
- MARQUES, A.S.A.; MARCHAISON, A.; GARDAN, L. & SAMSON, R. BOX-PCR-based identification of bacterial species belonging to Pseudomonas syringae - P. viridiflava group. Gen. Molec. Biol., 31:106-115, 2008.
- McCAIG, A.E.; GLOVER, L.A. & PROSSER, J.I. Numerical analysis of grassland bacterial community structure under different land management regimes by using 16S ribosomal DNA sequence data and denaturing gradient gel electrophoresis banding patterns. Appl. Environ. Microbiol., 67:4554-4559, 2001.
- MEDEIROS, A.F.A.; POLIDORO, J.C. & REIS, V.M. Nitrogen source effect on Gluconacetobacter diazotrophicus colonization of sugarcane (Saccharum spp.). Plant Soil, 279:141-152, 2006.
- MENDES, R.; KRUIJT, M.; BRUIJN, I.; DEKKERS, E.; van der VOORT, M.; SCHNEIDER, J.H.M.; PICENO, Y.M.; DE SANTIS, T.D.; ANDERSEN, G.L.; BAKKER, P.A.H.M. & RAAIJMAKERS, J.M. Deciphering the rhizosphere microbiome for disease-suppressive bacteria. Science, 332:1097-1100, 2011.
- MENDES, R.; PIZZIRANI-KLEINER, A.A.; ARAUJO, W.L. & RAAIJMAKERS, J.M. Diversity of cultivated endophytic bacteria from sugarcane: Genetic and biochemical characterization of Burkholderia cepacia complex isolates. Appl. Environ. Microbiol., 73:7259-7267, 2007.
- NASCIMENTO, C.B. Isolamento de microrganismos endofíticos da rizosfera de cana-de-açúcar (Saccharum officinarum L.) e análise da diversidade genética, antimicrobiana e celulolítica. R. Biol. Neotr., 3:187-188, 2006.
- NÜSSLEIN, K. & TIEDJE, J.M. Soil bacterial community shift correlated with change from forest to pasture vegetation in a tropical soil. Appl. Environ. Microbiol., 65:3622-3626, 1999.
- PATRIQUIN, D.G. & DÖBEREINER, J. Light microscopy observations of tetrazolium-reducing bacteria in the endorhizosphere of maize and other grasses in Brazil. Can J. Microbiol., 24:734-742, 1978.
- PHILIPPOT, L.; RAAIJMAKERS, J.; LEMANCEAU, P. & van der PUTTEN, W.H. Going back to the roots: The microbial ecology of the rhizosphere. Nature R. Microbiol., 11:789-799, 2013.
- SOGIN, M.L.; MORRISON, H.G.; HUBER, J.A.; WELCH, D.M.; HUSE, S.M.; NEAL, P.R.; ARRIETA, J.M. & HERNDL, G.J. Microbial diversity in the deep sea and the underexplored "rare biosphere". Proc. National Acad. Sci. USA, 103:12115-12120, 2006.
- VISUALIZATION AND ANALYSIS OF MICROBIAL POPULATION STRUCTURES - VAMPS. 2012. Disponível em: <http://vamps.mbl.edu/resources/>. Acesso em: 10 jun. 2012.
» link
Datas de Publicação
-
Publicação nesta coleção
30 Jan 2015 -
Data do Fascículo
Dez 2014
Histórico
-
Recebido
20 Fev 2014 -
Aceito
05 Set 2014